※온라인 안심존 : 보안이 보장된 온라인 네트워크를 통해 집, 연구실, 사무실 등 어디서나 접속하여 데이터에 접근하고 분석
-
데이터 변경이력
데이터 변경이력 버전 일자 변경내용 비고 1.0 2022-07-29 데이터 최초 개방 데이터 히스토리
데이터 히스토리 일자 변경내용 비고 2022-07-29 콘텐츠 최초 등록 소개
위장관 내시경 검사에서 채취된 조직의 병리검사 이미지 12,006장으로 구성된 데이터셋
구축목적
위장관내시경 조직 이미지를 판독하여 병리 진단을 하기 위한 데이터셋
-
메타데이터 구조표 데이터 영역 헬스케어 데이터 유형 이미지 데이터 형식 dcm, jpg 데이터 출처 자체수집 라벨링 유형 바운딩박스, 세그멘테이션, 텍스트 라벨링 형식 json, csv 데이터 활용 서비스 전문 병리 의사의 업무 보조 및 새로운 바이오마커 탐색 데이터 구축년도/
데이터 구축량2021년/12,006건 -
1. 데이터 구축 규모
1. 데이터 구축 규모 분류 진단명 (시험군의 경우 수집조건) 증례수 위 gastric adenocarcinoma 1,102 gastric adenoma 603 Gastritis with atypia 351 gastritis with intestinal metaplasia 350 normal stomach 1,100 H.pylori evaluation group (WSI수=700) 350 대장 intestinal adenocarcinoma 600 intestinal conventional adenoma 1,100 intestinal serrated lesion 550 Inflammatory bowel disease 350 intestinal polypoid lesion 350 normal intestine 1,100 위, 대장 gastric lymphoma, intestinal lymphoma 550 gastric neuroendocrine neoplasm, 550 gastric non-epithelial neoplasm other than lymphoma, intestinal neuroendocrine neoplasm, intestinal non-epithelial neoplasm other than lymphoma 시험군 일정 기간 내시경 생검 전체 증례 3,000 2. 데이터 분포
- 연령 분포
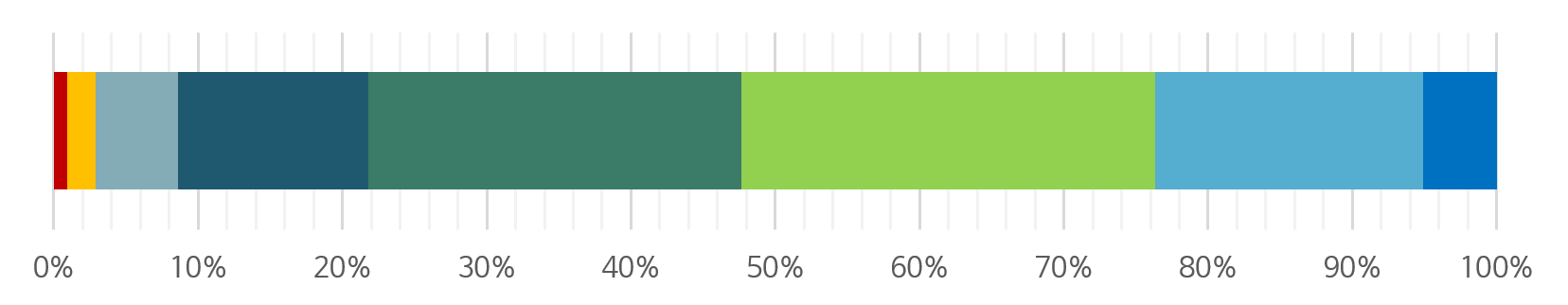
색상 연령대 건수 비율 10대 이하 113 0.94% 20대 235 1.96% 30대 682 5.68% 40대 1590 13.24% 50대 3104 25.85% 60대 3437 28.63% 70대 2232 18.59% 80대 이상 613 5.11% - 성별 분포
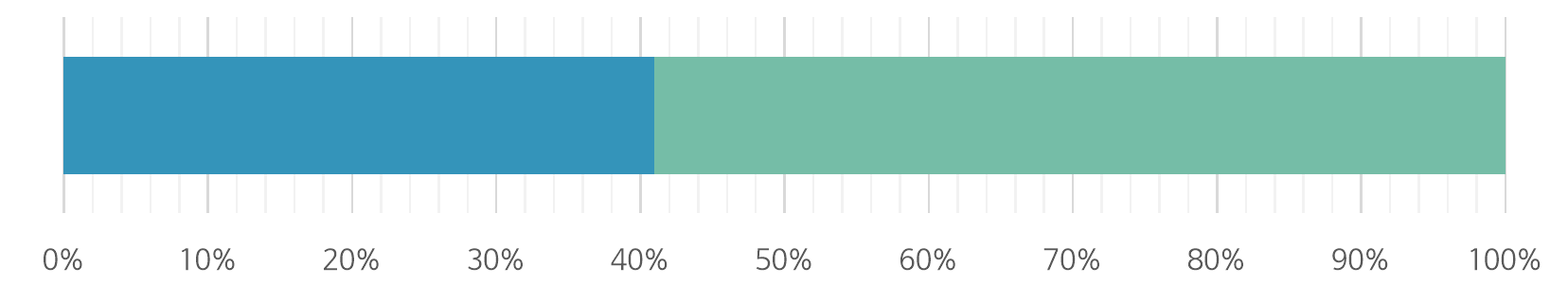
색상 성별 건수 비율 여성 4921 40.99% 남성 7085 59.01% - 장기 분포
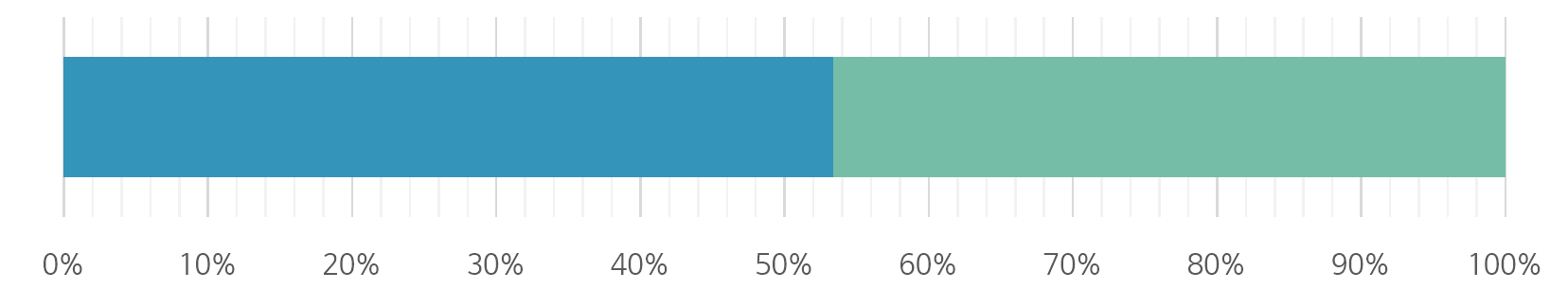
색상 장기 건수 비율 위 6413 53.42% 대장 5593 46.59% - 검체종류 분포
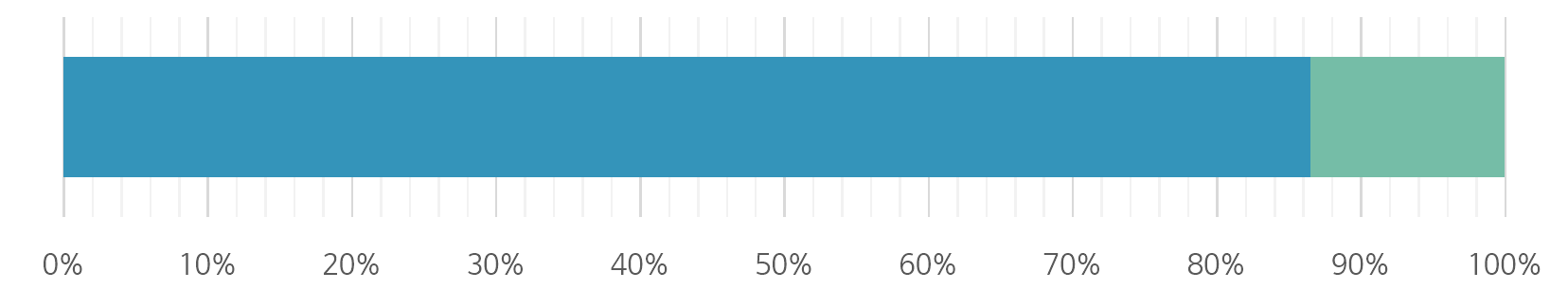
색상 검체종류 건수 비율 endoscopic biopsy 10390 86.54% endoscopic resection 1616 13.46% - 진단별 분포
- 장기: 위
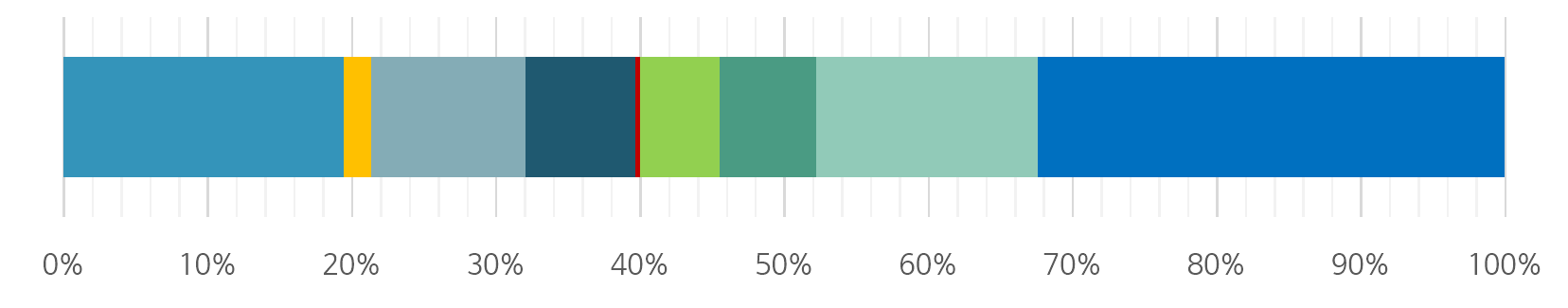
장기 색상 진단명 수량 백분율 위 gastric adenocarcinoma 1244 19.48% gastric neuroendocrine neoplasm 121 1.90% gastric adenoma 685 10.73% gastric lymphoma 483 7.56% gastric non-epithelial neoplasm other than lymphoma
(melanoma, GIST, leiomyoma, lipoma)24 0.38% H.pylori evaluation group 350 5.48% gastritis with atypia 431 6.75% gastritis with intestinal metaplasia 980 15.35% normal stomach 2067 32.37% 합계 6385 100% - 장기: 대장
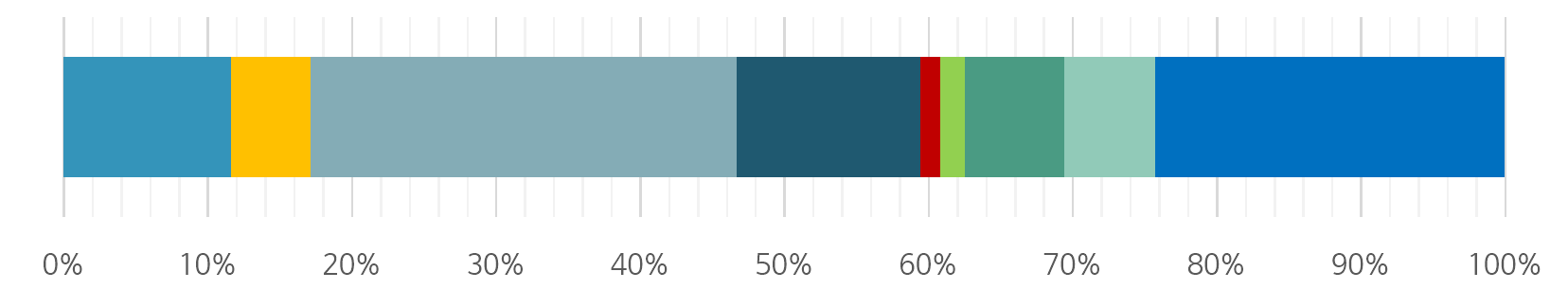
장기 색상 진단명 수량 백분율 대장 intestinal adenocarcinoma 655 11.65% intestinal neuroendocrine neoplasm 309 5.50% intestinal conventional adenoma 1661 29.55% (including tubular, villous, tubulovillous) intestinal serrated lesion (including HP, SSL, TSA) 718 12.77% intestinal lymphoma 77 1.37% ntestinal non-epithelial neoplasm other than lymphoma
(melanoma, GIST, leiomyoma, lipoma)97 1.73% Inflammatory bowel disease (including UC and CD) 388 6.90% intestinal polypoid lesion (including hamartomatous 355 6.32% polyp, inflammatory polyp, and other polypoid lesion) normal intestine 1361 24.21% 합계 5621 100%
- 장기: 위
- 연령 분포
-
-
AI 모델 상세 설명서 다운로드
AI 모델 상세 설명서 다운로드 AI 모델 다운로드1. 활용 모델
모델 학습- 대장암 : 대장암 검출 학습 모델
- 1 단계: Multi-Resolution Input CNN (Patch-level classifier) -> High resolution input patch와 low resolution input patch를 분석에 같이 사용하여 더 강건하고 일반적인 성능의 모델 개발 시도
- High Resolution Input: 분석을 실행하고자 하는 대상이며 동시에 조직 이미지 분석에 있어서 세포 핵의 크기나 모양과 같은 국소적 시각 정보 (Local Visual Information) 를 주로 추출하는 입력 데이터
- Low Resolution Input: High Resolution Input의 분석을 돕고자 해당 조직 영역의 주변의 전역적 문맥 정보 (Global Context Information) 를 주로 추출하는 입력 데이터
- 2 단계: 앙상블 모델 (Slide-level classifier)
- 학습 완료된 1단계 모델을 이용하여 WSI의 여러 특징들을 추출하고 해당 특징을 앙상블 모델로 학습 시켜 WSI 단위의 분류기 모델을 학습 시킴.
- 학습 방법:
- 데이터 분배 : Train : Validation : Test = 8 : 1 : 1
- 모델 parameters:
- batch size: 256
- optimizer: adamW
- initial learning rate: 0.001
- schedulear: cosine annealing warm restarts
- max epoch: 150
- weight decay: 1e-5
- 1 단계: Multi-Resolution Input CNN (Patch-level classifier) -> High resolution input patch와 low resolution input patch를 분석에 같이 사용하여 더 강건하고 일반적인 성능의 모델 개발 시도
- 위암 : 위암 검출 학습 모델
- 데이터 전처리
- 학습용 패치 추출
- Training set, Validation set에 있는 슬라이드의 mpp를 0.51로 표준화 이후 패치 추출
- Positive annotation annotation에서 패치 추출
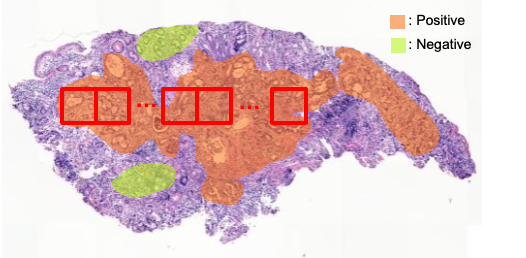
[학습 패치 추출 예시]
- 인공지능 모델 학습
- 알고리즘 설계
- 높은 해상도를 유지하기 위해 patch level의 학습 진행
- Slide 당 patch의 tumor probability의 평균값으로 slide의 tumor probability를 예측
- Tumor probability가 0.5 이상 일시 위암 진단, 아닐 시 정상 진단
- 사용 모델
- EfficientNet-B0 : 모델의 스케일 (width, depth, resolution)을 최적화 하여 가벼우면서 높은 정확도 때문에 현재 가장 널리 쓰이는 모델 (논문 인용 수 4,630회)
- ResNet50 : Residual block을 통해 모델 성능을 크게 끌어올려 전통적으로 가장 많이 사용하는 모델 (논문 인용 수 99,816회)
- 학습 환경
- Program language: python 3.8.10
- Framework: pytorch 1.9
- Patch size: 256
- Optimizer: Adam
- Learning rate scheduler: ReduceLROnPlateau
- Epoch: 100
- Batch size: [64, 128, 256]
- Initial learning rate: [1e-4, 1e-5, 1e-6]
- 알고리즘 설계
- 제언
- 위에서 제시하는 학습 환경은 최적화된 옵션이 아닌, 본 데이터를 활용해 모델 개발을 하기 위한 최소한의 가이드라인임
- 데이터 전처리
2. 서비스 활용 시나리오
- Slide level malignant tumor screening : 주어진 소화기 병리조직 WSI의 malignant tumor 유무를 판별하여 빠르게 slide screening을 진행하고, tumor 영역의 시각화를 통해 전문 병리 의사의 업무를 보조
- tumor visualization : 주어진 소화기 병리조직 WSI내에서 malignant tumor의 위치 영역을 시각화하여 전문 병리 의사의 진단 보조
- AI 추가 판독 (Second read) service : 증가하고 있는 판독 건수 대비 부족한 병리 전문의의 수를 고려할 때, 병리 의사 한 명이 부담해야 하는 업무량과 피로가 증가하고 있음. AI 기반 추가 판독을 통해 의료 사고 (e.g. Tumor를 놓치는 케이스)에 대한 안전 기준을 높일 수 있음
- 대장암 : 대장암 검출 학습 모델
-
데이터 성능 점수
측정값 (%)기준값 (%)데이터 성능 지표
데이터 성능 지표 번호 측정항목 AI TASK 학습모델 지표명 기준값 점수 측정값 점수 1 위암 진단 분류 모델 Image Classification EfficientNet-B0 기반 CNN 모델 AUC-ROC 0.7 단위없음 0.9533 단위없음 2 대장암 진단 분류 모델 Image Classification Multi-Resolution Input CNN AUC-ROC 0.7 단위없음 1 단위없음
※ 데이터 성능 지표가 여러 개일 경우 각 항목을 클릭하면 해당 지표의 값이 그래프에 표기됩니다.
※ AI모델 평가 지표에 따라 측정값의 범위, 판단 기준이 달라질 수 있습니다. (ex. 오류율의 경우, 낮을수록 좋은 성능을 내는 것으로 평가됩니다)
-
설명서 및 활용가이드 다운로드
데이터 설명서 다운로드 구축활용가이드 다운로드1. 대표도면
- multiframe이미지 : dcm
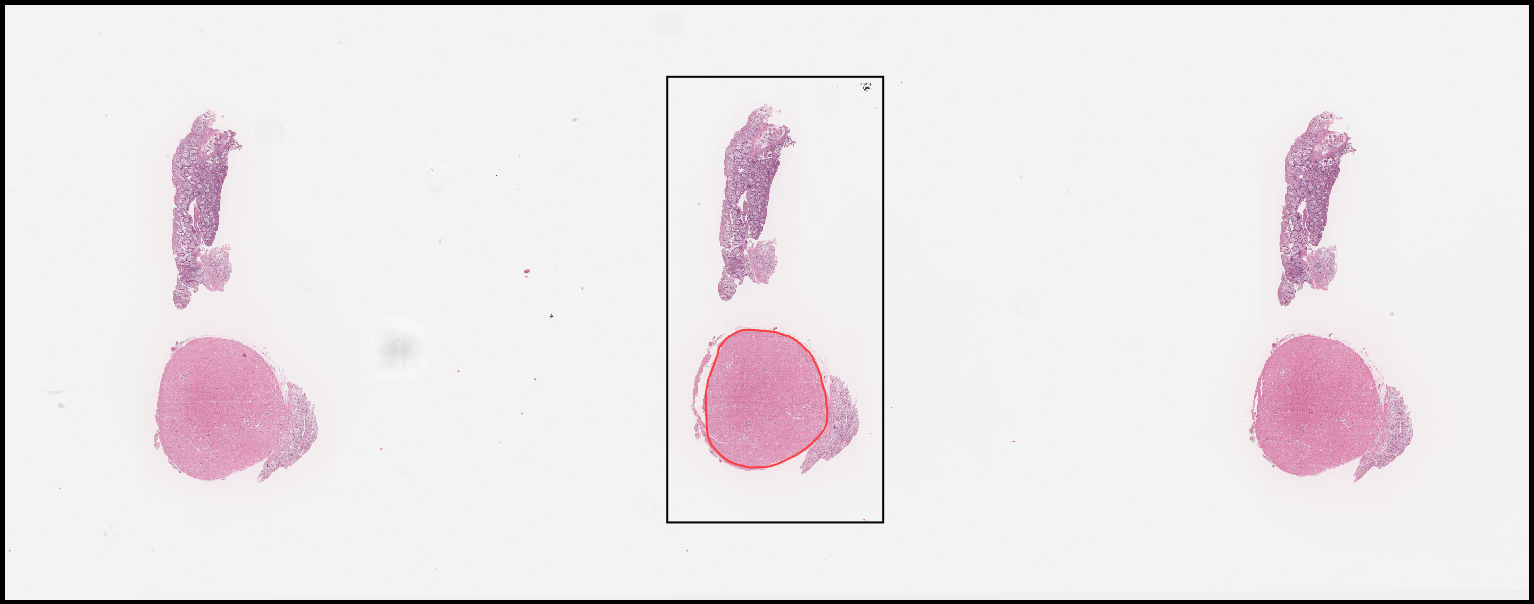 [어노테이션 정보가 포함된 multiframe이미지의 예시]
[어노테이션 정보가 포함된 multiframe이미지의 예시] - single frame이미지 : jpg
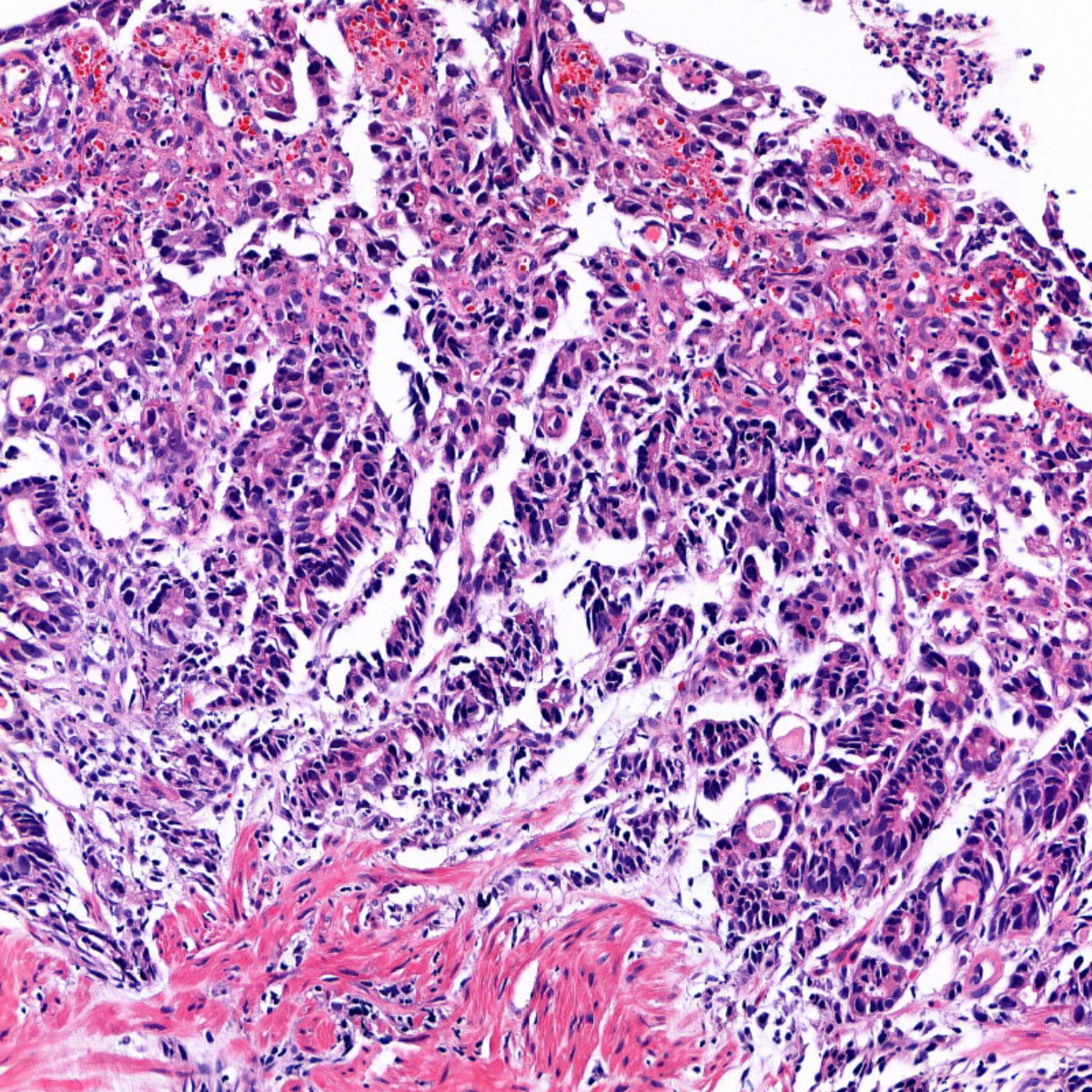
2. 라벨링데이터 구성 (필수 : M / 필수아님 : O)
- 진단 라벨 : 1.Dataset>원천데이터>(case_id)>(case_id).csv
1. 대표도면 구분 항목명 타입 필수여부 설명 범위 참조 1 case_id string M 증례 일련번호 NA 2 slide_id string M 이미지원천데이터 일련번호 NA 3 age int M 진단시 연령 [0,200] 붙임1 4 sex int M 성별 0 or 1 붙임1 5 los int M 장기 0 or 1 붙임1 6 los01 int O 위장 세부위치 [0,3] 붙임1 7 los11 int O 대장 세부위치 [0,6] 붙임1 8 proc int M 검체종류 0 or 1 붙임1 9 dx_qc int M 진단 [0,17] 붙임1 10 dx_code string M 세부진단 코드
(code for diagnosis)NA 붙임1 11 tstg0 int O 위암병기 T category [0,6] 붙임1 12 nstg0 int O 위암병기 N category [0,4] 붙임1 13 mstg0 int O 위암병기 M category 0 or 1 붙임1 14 tstg1 int O 대장암병기 T category [0,5] 붙임1 15 nstg1 int O 대장암병기 N category [0,5] 붙임1 16 mstg1 int O 대장암병기 M category [0,4] 붙임1 17 preop int O 수술전 항암치료 0 or 1 붙임1 18 msi0 int O MSI검사결과 0 or 1 붙임1 19 ebv0 int O EBV검사결과 0 or 1 붙임1 20 her20 int O HER2검사결과 0 or 1 붙임1 21 kras1 int O KRAS검사결과 0 or 1 붙임1 22 nras1 int O NRAS검사결과 0 or 1 붙임1 23 braf1 int O BRAF검사결과 0 or 1 붙임1
[붙임1]항목명 비고 age sex 0=female, 1=male los 0=stomach, 1=large intestine los01 0=antrum, 1=angle, 2=body 3=cardia/EJG, 9999=NA los11 0=cecum, 1=ascending colon, 2=transverse colon, 3=descending colon, 4=sigmoid, 5=rectosigmoid, 6=rectum, 9999=NA proc 0=biopsy, 1=resection tstg0 0=Tis, 1=T1a, 2=T1b, 3=T2, 4=T3, 5=T4a, 6=T4b, 9999=NA nstg0 0=N0, 1=N1, 2=N2, 3=N3a, 4=N3b, 9999=NA mstg0 0=M0, 1=M1, 9999=NA tstg1 0=Tis, 1=T1, 2=T2, 3=T3, 4=T4a, 5=T4b, 9999=NA nstg1 0=N0, 1=N1a, 2=N1b, 3=N1c, 4=N2a, 5=N2b, 9999=NA mstg1 0=M0, 1=M1, 9999=NA preop 0=no, 1=yes msi0 0=intact, 1=abnormal, 9999=NA ebv0 0=negative, 1=positive, 9999=NA her20 0=not amplified, 1=amplified, 9999=NA kras1 0=wild, 1=mutant, 9999=NA nras1 0=wild, 1=mutant, 9999=NA braf1 0=wild, 1=mutant, 9999=NA dx_qc 0=gastric adenocarcinoma, 1=gastric neuroendocrine neoplasm, 2=gastric adenoma, 3=gastric lymphoma, 4=gastric non-epithelial neoplasm other than lymphoma (melanoma, GIST, leiomyoma, lipoma), 5=H.pylori evaluation group, 6=gastritis with atypia, 7=gastritis with intestinal metaplasia, 8=normal stomach, 9=intestinal adenocarcinoma, 10=intestinal neuroendocrine neoplasm, 11=intestinal conventional adenoma (including tubular, villous, tubulovillous), 12=intestinal serrated lesion (including HP, SSL, TSA), 13=intestinal lymphoma, 14=intestinal non-epithelial neoplasm other than lymphoma (melanoma, GIST, leiomyoma, lipoma), 15=Inflammatory bowel disease (including UC and CD), 16=intestinal polypoid lesion (including hamartomatous polyp, inflammatory polyp, and other polypoid lesion), 17=normal intestine dx_detail 세부진단 code for diagnosis Stomach Papillary adenocarcinoma c0000001 Stomach Tubular adenocarcinoma WD c0000002 Stomach Tubular adenocarcinoma MD c0000003 Stomach Tubular adenocarcinoma PD c0000014 Stomach Poorly cohesive carcinoma c0000015 Stomach Signet ring cell carcinoma c0000016 Stomach Mucinous adenocarcinoma c0000017 Stomach Medullary carcinoma with lymphoid stroma c0000018 Stomach Tubular adenoma high grade c0001100 Stomach Tubular adenoma low grade c0001110 Stomach Atypia vienna 2 c0110000 Stomach Atypia vienna 1 c0110100 Stomach Neuroendocrine Neoplasm c0000100 Stomach H.pylori positive c0100000 Stomach H.pylori negative c0100100 Stomach Intestinal metaplasia c0111000 Stomach Normal c0111100 Stomach Lymphoma c0010000 Stomach Melanoma c0010100 Stomach Gastrointestinal stromal tumor c0012100 Stomach Leiomyoma c0011120 Stomach Lipoma c0011130 Intestine Adenocarcinoma WD c1000000 Intestine Adenocarcinoma MD c1000010 Intestine Adenocarcinoma PD c1000020 Intestine Neuroendocrine Neoplasm c1000100 Intestine Tubular adenoma high grade c1001000 Intestine Tubular adenoma low grade c1001001 Intestine Tubulovillous adenoma high grade c1001010 Intestine Tubulovillous adenoma low grade c1001011 Intestine Villous adenoma high grade c1001020 Intestine Villous adenoma low grade c1001021 Intestine Hyperplastic polyp c1001100 Intestine Sessile serrated lesion c1001101 Intestine Traditional serrated adenoma c1001102 Intestine Lymphoma c1010000 Intestine Melanoma c1010100 Intestine Gastrointestinal stromal tumor c1012000 Intestine Leiomyoma c1011000 Intestine Lipoma c1011100 Intestine Crohndisease c1100000 Intestine Ulcerative colitis c1101000 Intestine Hamartomatous polyp c1110000 Intestine Inflammatory polyp c1110100 Intestine Other polypoid lesion c1110200 Intestine Normal c1111000 - 이미지 영역 라벨 : 1.Dataset>라벨링데이터>(case_id)>(slide_id).json
구분 항목명 타입 필수여부 설명 범위 1 type string M Annotation의 타입 “Feature” 2 geometry object M 영역을 정의하는 객체 NA 2-1 type string M Geometry의 타입 “Polygon”, “Bounding box” 2-2 coordinates object M 좌표 객체 NA 2-2-1 [] array M 포인트 배열 NA 2-2-1-1 [] array M 포인트 성분 배열 NA 2-2-1-1-1 number M 원소 1 : column 0 < 원소 2 : row 3 Properties object M 속성을 정의하는 객체 3-1 object_type string M 객체의 타입 “annotation” 3-2 classification object M Annotation 분류 객체 NA 3-3 name string M 분류 명 “Region*”, “Positive”, “Negative” 3-4 colorRGB number M Annotation Line 색상 3-5 isLocked boolean O Annotation 수정 가능 여부 ture, false 3-6 measurements [] array O 영역 측정 정보 배열 NA 3-6-1 {} object O 측정 항목 객체 NA 3-6-1-1 name string O 측정 항목 명 “Area”, “Perimeter” 3-6-1-2 value number O 측정 항목 값
3. 라벨링데이터 실제예시
- 진단데이터 : csv

- 어노테이션 데이터 : json


- multiframe이미지 : dcm
-
데이터셋 구축 담당자
수행기관(주관) : 소화기병리학연구회/국립암센터
수행기관(주관) 책임자명 전화번호 대표이메일 담당업무 국명철 031-920-1745 mckook@ncc.re.kr · 데이터 구축작업 총괄 · 데이터 설계 · 데이터 수집-정제-가공-검수 수행기관(참여)
수행기관(참여) 기관명 담당업무 가톨릭대학교 산학협력단 서울성모병원 · 데이터 수집-정제-가공-검수 서울대학교 산학협력단 · 데이터 수집-정제-가공-검수 연세대학교 산학협력단 · 데이터 수집-정제-가공-검수 연세대학교 원주산학협력단 · 데이터 수집-정제-가공-검수 녹십자의료재단 · 데이터 수집-정제-가공-검수 인피니트헬스케어 · 저작도구 개발
· 데이터 정제-가공(주) 뷰노 · AI모델 개발 에이아이트릭스 · AI모델 개발 웹프라임 · 개발환경 및 공동작업환경 구축
· 데이터 저장소 관리데이터 관련 문의처
데이터 관련 문의처 담당자명 전화번호 이메일 국명철 031-920-1745 mckook@ncc.re.kr
-
인터넷과 물리적으로 분리된 온라인·오프라인 공간으로 의료 데이터를 포함하여 보안 조치가 요구되는 데이터를 다운로드 없이 접근하고 분석 가능
* 온라인 안심존 : 보안이 보장된 온라인 네트워크를 통해 집, 연구실, 사무실 등 어디서나 접속하여 데이터에 접근하고 분석
* 오프라인 안심존 : 추가적인 보안이 필요한 데이터를 대상으로 지정된 물리적 공간에서만 접속하여 데이터에 접근하고 분석 -
- AI 허브 접속
신청자 - 안심존
사용신청신청자신청서류 제출* - 심사구축기관
- 승인구축기관
- 데이터 분석 활용신청자
- 분석모델반출신청자
- AI 허브 접속
-
1. 기관생명윤리위원회(IRB) 심의 결과 통지서 [IRB 알아보기]
2. 기관생명윤리위원회(IRB) 승인된 연구계획서
3. 신청자 소속 증빙 서류 (재직증명서, 재학증명서, 근로계약서 등 택1)
4. 안심존 이용 신청서 [다운로드]
5. 보안서약서 [다운로드]
※ 상기 신청서 및 첨부 서류를 완비한 후 신청을 진행하셔야 정상적으로 절차가 이루어집니다. -
신청 및 이용관련 문의는 safezone1@aihub.kr 또는 02-525-7708, 7709로 문의
데이터셋 다운로드 승인이 완료 된 후 API 다운로드 서비스를 이용하실 수 있습니다.
API 다운로드 파일은 분할 압축되어 다운로드 됩니다. 분할 압축 해제를 위해서는 분할 압축 파일들의 병합이 필요하며 리눅스 명령어 사용이 필요합니다.
리눅스 OS 계열에서 다운로드 받으시길 권장하며 윈도우에서 파일 다운로드 시 wsl(리눅스용 윈도우 하위 시스템) 설치가 필요합니다.
※ 파일 병합 리눅스 명령어
find "폴더경로" -name "파일명.zip.part*" -print0 | sort -zt'.' -k2V | xargs -0 cat > "파일명.zip"
- 해당 명령어 실행 시, 실행 경로 위치에 병합 압축 파일이 생성됩니다.
- 병합된 파일 용량이 0일 경우, 제대로 병합이 되지 않은 상태이니 "폴더경로"가 제대로 입력되었는지 확인 부탁드립니다.
※ 데이터셋 소개 페이지에서 다운로드 버튼 클릭하여 승인이 필요합니다.